Dr. Alexander Jarasch, Dr. Astrid Glaser,
Prof. Dr. Martin Hrabě de Angelis
Vernetzte Daten für eine Diabetesforschung der Zukunft
Die Menge an Daten zum Thema Diabetes wächst kontinuierlich. Die Volkskrankheit Diabetes lässt sich in einer vollkommen neuen Dimension erforschen – wenn es gelingt, die vielfältigen und bisher meist in separaten Datenbanken gespeicherten Informationen miteinander zu vernetzen und mit modernen Informations- und Kommunikationstechnologien wie der künstlichen Intelligenz zu analysieren.
Bei Hausärzten, in Kliniken, in Universitäten, Forschungseinrichtungen, Labors, bei den Krankenkassen, in Pharmaunternehmen: Die Menge an medizinischen Daten wächst kontinuierlich und exponentiell. Dabei reicht die Datenvielfalt von Blutwerten, Biomarkern, Befunden, Ultraschallaufnahmen sowie Röntgen- und CT-Bildern bis in das menschliche Genom. Hinzu kommen unzählige Daten aus Studien und Publikationen sowie Patientendaten, die heute nicht mehr nur beim Arzt gesammelt werden, sondern auch rund um die Uhr von medizinischen Geräten, Gesundheits- oder Sport-Apps. Allein in den USA umfasst die Menge der Gesundheitsdaten bereits mehr als 150 Exabyte – das sind 150 Mio. Terabyte [Raghupathi 2014].
In diesen Datenbergen schlummern ungeheure Schätze. Doch „Big Data“ bietet nicht per se einen Mehrwert. Um die verborgenen Schätze bergen zu können, müssen die Daten auch zugänglich, interoperabel und durchsuchbar sein. Erst dann lässt sich durch den Einsatz moderner IT-Lösungen wie Data Mining und künstlicher Intelligenz (siehe „Definitionen“) „Big Data“ in „Smart Data“ verwandeln.
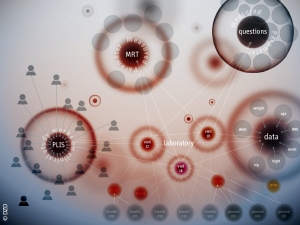
In der Diabetesforschung gibt es derzeit vielfältige separate Daten
Das gilt insbesondere auch für die Volkskrankheit Diabetes. Diabetes ist eine äußerst komplexe Erkrankung, die durch ein vielschichtiges Zusammenspiel von Genen, Lebensstil und Umweltfaktoren entsteht. Daher erforschen zahlreiche Arbeitsgruppen und Experten verschiedener Disziplinen (u. a. aus Grundlagenforschung, Epidemiologie, Versorgungsforschung und Klinik) die Stoffwechselerkrankung. Jede dieser Gruppen sammelt jeweils wichtige Daten zu einzelnen Aspekten der Erkrankung. In der Diabetesforschung stammen die Daten u. a. aus Kohorten, klinischen Studien, Bioproben, präklinischen Modellen, Geno- und Phäntypisierung, Omics-Analysen sowie aus Untersuchungen zu epigenetischen Veränderungen des genetischen Codes. Die Daten und Ergebnisse der Studien und Untersuchungen sind jedoch sehr heterogen, oft unstrukturiert und werden an verschiedenen Standorten in separaten Datenbanken (Datensilos) gespeichert. Zudem sind die Informationen meist gar nicht oder nur sehr schwer durchsuchbar. Doch um die Entstehung der Krankheit zu verstehen und neue präzise Therapieansätze (Precision Medicine) entwickeln zu können, müssen Informationen aus unterschiedlichen Bereichen miteinander verbunden und ausgewertet werden. Denn mehr als jede einzelne Datenquelle kann die Vernetzung aller verfügbaren Datenbanken das Wissen zur Diabetesentstehung, -prävention und -behandlung erweitern.
Forschungsdaten miteinander vernetzen
Das Deutsche Zentrum für Diabetesforschung (DZD) – mit fünf Partnern und fünf assoziierten Partnern – entschloss sich daher, DZDconnect als standortübergreifendes Daten- und Knowledge-Management aufzubauen. Die Grundlage dafür bildet eine Graphdatenbank. Sie soll die bereits vorhandenen Daten aus unterschiedlichen Quellen im DZD zugänglich, wiederverwertbar und übergreifend nutzbar machen – gemäß dem FAIR-Prinzip (Findable, Accessible, Interoperable und Re-usable). Damit will das DZD in Zukunft seine Forschungsdaten aus den heterogenen Quellen zusammenführen, strukturieren und mithilfe innovativer IT-Technologien – wie Artificial Intelligence – analysieren, um so bisher unerkannte Querverbindungen und Muster sichtbar zu machen [Möller 2019, Jarasch 2018]. Im Sinne eines interdisziplinären Forschungsansatzes lassen sich so bislang verborgene Zusammenhänge der Krankheit aufdecken. Warum erkranken manche Menschen an Diabetes, andere nicht? Welche Rolle spielen dabei Gene, Ernährung, Bewegung und Umweltfaktoren? Lässt sich schon in jungen Jahren das Diabetesrisiko bestimmen? Und wie kann man der Erkrankung entsprechend vorbeugen? Die Verknüpfung unterschiedlicher Datensätze kann hier grundsätzlich neue Antworten liefern. So könnten z. B. neue Marker für Prädiabetes oder andere Subtypen des Diabetes entdeckt werden. Zudem könnte das System über Natural Language Processing (NLP) in Zukunft Texte einlesen, analysieren und relevante Informationen selbstständig in die Datenbank integrieren, sobald zentrale Begriffe signifikant häufig oder im richtigen semantischen Umfeld auftreten.
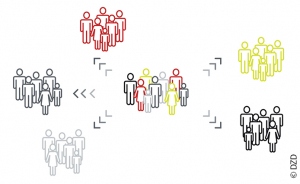
Subtypen des Diabetes entdeckt
Diabetes ist eine Erkrankung, die sich sehr heterogen manifestiert. Studien aus Skandinavien [Ahlqvist 2018] zeigten, dass man Diabetiker entsprechend der Schwere des Verlaufs des Diabetes in verschiedene Cluster einteilen kann. Das DZD konnte diese Befunde an 1 105 Patienten aus der Deutschen Diabetes-Studie bestätigen. Mithilfe der in Schweden erprobten Analysemethode (Clustering-Algorithmus) suchten die Wissenschaftler in dem umfangreichen Datensatz nach auffälligen Mustern und Gesetzmäßigkeiten und entdeckten fünf Cluster, die eine Aufteilung des Diabetes in Subtypen ermöglichen. Zwei dieser neuen Subtypen – der milde adipositasbedingte Diabetes und der milde altersbedingte Diabetes – haben weniger schwere Krankheitsverläufe. Die drei übrigen Subtypen gehen mit einem hohen Risiko von Diabetesfolgen einher: Patienten mit schwerem insulinresistentem Diabetes haben vermutlich ein höheres Risiko für Erkrankungen der Leber und Nieren, während jene mit einem schweren insulindefizitären Diabetes eher an Netzhautschäden und diabetischer Neuropathie erkranken dürfen. Der dritte Subtyp mit häufigen Komplikationen ist der schwere autoimmune Diabetes, der dem klassischen Typ-1-Diabetes entspricht [Zaharia 2019].
Unterschiedliche Arten des Prädiabetes
Unterschiedliche Subtypen gibt es bereits bei der Vorstufe des Diabetes (Prädiabetes). Das zeigen Auswertungen der DZD-Multicenterstudie „Prädiabetes-Lebensstil-Interventions-Studie“ (PLIS). Nicht jeder Prädiabetiker hat das gleich hohe Risiko, später auch einen Diabetes zu entwickeln. Es gibt vielmehr eine Hochrisikogruppe. Bei Probanden, die an einer Fettleber mit Insulinresistenz oder einer Insulinsekretionsstörung leiden, kommt es mit einer sehr hohen Wahrscheinlichkeit zu einer manifesten Diabeteserkrankung. Zudem ist das Risiko erhöht, später auch Folgeerkrankungen zu entwickeln. Untersuchungen deuten darauf hin, dass eine intensive Lebensstilintervention mit viel Bewegung und einer nachhaltig begleitenden Beratung hier helfen kann, den Ausbruch der Stoffwechselerkrankung hinauszuzögern oder gar zu verhindern [Fritsche 2019].
Digitales Diabetes Präventions-Zentrum (DDPC)
Wir müssen aber davon ausgehen, dass noch mehr als diese schon beschriebenen Subtypen des Diabetes bzw. Prädiabetes existieren. Daher gilt es, weitere Cluster – respektive Patientengruppen – zu identifizieren und herauszufinden, welche davon jeweils welche Eigenschaften aufweisen und wie die verschiedenen Subtypen am besten erkannt und behandelt werden könnten. Doch um diese Untergruppen entdecken zu können, bedarf es sehr viel größerer Datenmengen. Daher wollen wir ein Digitales Diabetes Präventions-Zentrum (DDPC) aufbauen. Der Datenpool für das DDPC soll sich nicht nur aus Forschungsdaten des DZD speisen, sondern auch aus weiteren vorhandenen medizinischen Daten sowie Gesundheitsdaten. Informationen, die bisher bei nationalen Forschungseinrichtungen sowie Krankenkassen, Krankenhäusern und niedergelassenen Ärzten vorliegen, können wertvolle Hinweise liefern. Zusätzlich werden Daten gesunder Personen aus der allgemeinen Bevölkerung gebraucht, um die Entstehung von Typ-2-Diabetes besser verstehen und die dringend benötigten frühen Prädiktoren für unterschiedliche Subtypen aufspüren zu können.
Mögliche Quellen für diese Informationen könnten die heute schon weitverbreiteten Tracking-Armbänder, smarten Uhren, Wearables oder Fitness- und Gesundheits-Apps sein. Bislang greifen vor allem große internationale IT- und Pharmaunternehmen auf selbst erhobene Gesundheitsdaten von Privatpersonen zu. Um diesen Datenschatz nicht vorrangig der Industrie zur Nutzung zu überlassen, ist es wichtig, dass auch die Forschung Zugang zu diesen Daten bekommt. Dem Wunsch, diese vielfältigen Datenquellen nutzen zu können, stehen derzeit strenge Datenschutzbestimmungen entgegen. Gemeinsam mit der Politik, den Fachgesellschaften und Patientenverbänden müssen wir deshalb nach Lösungen suchen, die den Missbrauch digitaler Daten ausschließen und zugleich deren Nutzung für die medizinische Forschung nicht behindern. Der von der Deutschen Diabetes Gesellschaft (DDG) entwickelte „Code of Conduct Digital Health“ bietet hierzu eine gute Grundlage [Deutsche Diabetes Gesellschaft 2018].
Deutliche Mehrheit der Deutschen bereit zur Datenspende
Die Bereitschaft der Menschen, ihre Gesundheitsdaten in Form einer Datenspende der Wissenschaft zur Verfügung zu stellen, ist sehr groß. Fast vier von fünf Deutschen (79 %) würden ihre Gesundheitsdaten anonym und unentgeltlich digital für die medizinische Forschung zur Verfügung stellen. Das zeigt eine Forsa-Umfrage im Auftrag der Technologie- und Methodenplattform für die vernetzte medizinische Forschung [TMF 2019]. Ein ähnliches Ergebnis ergab sich auch in der in diesem D.U.T. veröffentlichten Untersuchung, in der 69,7 % aller befragten Patienten ihre Daten anerkannten Institutionen zu wissenschaftlichen Zwecken überlassen würden.
DDPC: Politik überzeugen
Das DDPC ist als nationale Initiative mit verschiedenen Standorten in Deutschland angedacht. Die mathematische und informationstechnische Fachkompetenz bringt das Helmholtz Zentrum München ein, die klinische Fachkompetenz kommt vom DZD und weiteren nationalen Partnern an Kliniken und Forschungseinrichtungen. Mit neuen IT-Technologien aus dem Bereich Artificial Intelligence ist es möglich, die oben beschriebenen unterschiedlichen Datenquellen miteinander zu verbinden und auszuwerten. Wichtig dabei ist die enge Zusammenarbeit von Datenexperten mit all jenen Experten aus Forschung und Klinik. Im DDPC sollen die Diabetes-Data-Institute von Klinischen Zentren des DZD flankiert werden, die auch „Proof of principle“-Studien durchführen. Aus dem so generierten Wissen lassen sich präzise Präventionsstrategien ableiten und individuelle Empfehlungen für (noch) gesunde Bürgerinnen und Bürger abgeben. Noch ist das DDPC eine Zukunftsvision. Es gilt, die Politik zu überzeugen, in ein solches Zentrum zu investieren.
Künstliche Intelligenz in der Diabetesforschung
Weltweit nutzen Diabetesforscher die neuen Möglichkeiten moderner IT-Technologien. An welch unterschiedlichen Fragestellungen die Wissenschaftler arbeiten, zeigen ein paar Beispiele: Wissenschaftlern aus München und Dresden ist es gelungen, u. a. mithilfe von Deep-Learning-Methoden, den Risk Score für Typ-1-Diabetes deutlich zu verbessern. Der neue Risikotest bezieht bis zu 41 Typ-1-Diabetes-Risikogen-Regionen ein [Bonifacio 2018]. IBM forscht an einem KI-gestützten Screening-Tool, um Typ-1-Diabetes frühzeitig diagnostizieren zu können [Hu 2019].
Derzeit arbeitet das Pharmaunternehmen Roche mit IBM Watson Health an einem neuen datenbasierten Vorhersagemodell (mit Real World Data) für diabetesbedingte chronische Nierenerkrankungen [Ravizza 2019]. In einem Pilotprojekt aus München gelang es Computational-Biology-Experten, mit Techniken von Deep-Learning Retinopathien – eine häufige Folgeerscheinung des Diabetes – besser zu klassifizieren. Sie trainierten den Computer darauf, kranke und gesunde Augen zu erkennen und die Patienten entlang der Schwere und des Fortschreitens der Erkrankung einzuordnen [Eulenberg 2017]. Diese Strategie wird nun weiterverfolgt, um neue Therapien und individuelle Vorsorgekonzepte zu entwickeln. Auch andere Gruppen arbeiten an Systemen zum Netzhautscreening, um diabetische Veränderungen an der Netzhaut zu erkennen. Bereits 2018 hat die amerikanische Gesundheitsbehörde (Food and Drug Administration, FDA) ein erstes System zur Anwendung künstlicher Intelligenz zur Retinopathiediagnostik zugelassen [U.S. Food and Drug Administration 2018]. Als erste Diabetesklinik in Deutschland setzt das Diabetes Zentrum Mergentheim seit Ende 2018 künstliche Intelligenz für Untersuchungen auf diabetische Retinopathie ein.
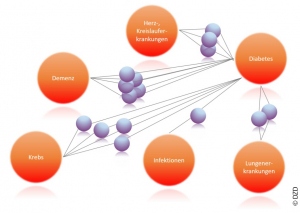
Verbindungen zwischen verschiedenen Volkskrankheiten erkennen
Diabetes ist mit vielen anderen großen Volkskrankheiten wie Demenz, Krebs, Lungen-, Infektions-, Herz- und Kreislauferkrankungen assoziiert. Um Zusammenhänge zwischen diesen Erkrankungen und mögliche gemeinsame frühe Prädiktoren zu erkennen, arbeiten die sechs Deutschen Zentren der Gesundheitsforschung (DZG) zusammen. Hier gilt es, Synergien zu nutzen und datenschutzkonforme Datenverknüpfungen aufzubauen.
Nationale Forschungsdateninfrastruktur
Um Daten sicher und datenschutzkonform forschungseinrichtungsübergreifend nutzen zu können, bedarf es entsprechender IT-Infrastrukturen. Ende 2018 haben Bund und Länder den Aufbau einer Nationalen Forschungsdateninfrastruktur (NFDI) beschlossen. In der NFDI sollen Datenbestände in einem aus der Wissenschaft getriebenen Prozess systematisch erschlossen, langfristig gesichert und entlang der FAIR-Prinzipien über Disziplinen- und Ländergrenzen hinaus zugänglich gemacht werden. Um den Aufbau dieser Infrastruktur konnten sich bis zum Herbst 2019 NFDI-Konsortien aller Fachrichtungen bewerben. Die DZG haben gemeinsam mit den vier Konsortien der Medizininformatik-Initiative (MII) unter der Federführung des DZD und der Technologie- und Methodenplattform für die vernetzte medizinische Forschung (TMF) einen Antrag für den Aufbau einer interoperablen, nutzerfreundlichen und nachhaltigen Infrastruktur für die Integration und den Austausch von Daten aus verschiedenen Bereichen der biomedizinischen Forschung und klinischen Versorgung gestellt. Ziel dieses neuen „NFDI for Future Medicine Konsortium (NFDI4MED)“ ist es, ein qualitätsorientiertes Datenmanagement als integralen Bestandteil des Forschungsdatenzyklus im Bereich der patientenorientierten biomedizinischen Forschung zu ermöglichen. Die Struktur des Forschungsdatenmanagements von NFDI4MED soll eine dezentrale nationale Infrastruktur umfassen, die vorhandene Datenbanken und digitale Ressourcen aus den verschiedenen Forschungsdisziplinen und -organisationen miteinander vernetzt. Im NFDI4Medicine-Konsortium sind dies mehr als 100 Universitäts- und Universitätsklinikstandorte, Forschungseinrichtungen und -organisationen sowie Bundesinstitute. Durch Maßnahmen wie diese wird ein wichtiger Grundstein gelegt, um die Menge an Daten, die in der biomedizinischen Forschung und klinischen Versorgung entstehen, für die Gesundheit der Menschen und neue Präventions- und Therapiemaßnahmen sinnvoll einzusetzen.




 AdobeStock - thodonal
AdobeStock - thodonal sturti - iStockphoto
sturti - iStockphoto